使用ACQUITY UPLC和Xevo TQ-S分析食品中致过敏物质肽
食品过敏情况越来越多,对食品呈不良反应的人口数量也一直在增加。尽管许多食品会导致发生反应,但90%的食品过敏反应局限于少量所谓的关键过敏原。这些关键过敏原包含在牛奶、甲壳动物、卵、鱼、花生、大豆、坚果和小麦中。为了帮助那些出现食品过敏的人,全球范围内都有与食品安全问题相关的许多不同法规。
许多年来,分析食品中是否出现过敏组分的最常见分析技术都基于酶联免疫吸附法和实时聚合酶链反应的抗体技术。ELISA基于可利用目标过敏蛋白质和不会引起其他类似蛋白质反应的抗体,PCR不会标记相应的特定目标蛋白质,因此会产生假阳性结果。由于能够标记特定蛋白质和一种方法分析多种过敏物质,最近使用质谱(MS)检测过敏原的方式引起很大关注。能够在方法开发过程中同时监测基质背景,优化出最佳方案,确保方法高效可靠。
该技术简介中,用于制作面包的花生被用于解释食品基质中的过敏原肽标记的检测方法。还介绍了在定量MRM分析过程中能够同时采集基质背景信息的仪器设计方面的重要进展。该功能(被称作RADARTM采集)为组分定量分析并且同时监测背景干扰提供了创新方法,能够在方法开发过程的同时持续监测基质背景,优化出最佳方案,确保方法高效可靠。
解决方案
之前的实验在沃特世(Waters®)nanoACQUITY UPLC®和Xevo QTof MS(应用序列号720003656EN)上使用食品蛋白质组学方法鉴定摄入生或烤花生引起的过敏反应相关标记,该实验鉴定了生和烤花生的肽序列。在本次实验中,使用其中两种肽段和ACQUITY UPLC系统和Xevo TQ-S质谱开发高度灵敏和高特异性的方法。
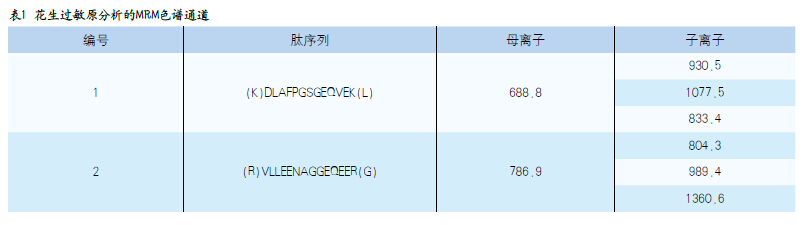
选择面包作为食品基质,花生蛋白质Ara h1作为标记过敏蛋白质,提取蛋白质后用胰蛋白酶消化,将选择的标记肽加入UPLC®/MS/MS方法中,使用Xevo TQ-S在RADAR模式下分析加标以及未加标面包基质。表1列出了花生中Ara h1的每种酶所选择的3种MRM色谱通道。图1下方显示了MRM总离子流图,插图显示了肽序列VLLEENAGGEQEER的三种提取离子MRM色谱图。
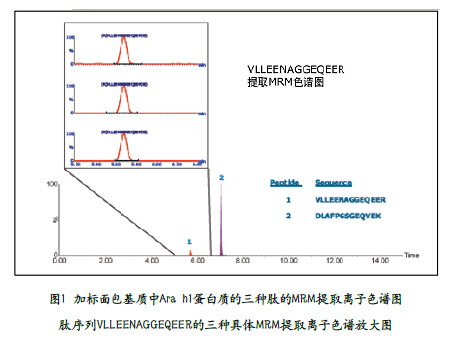
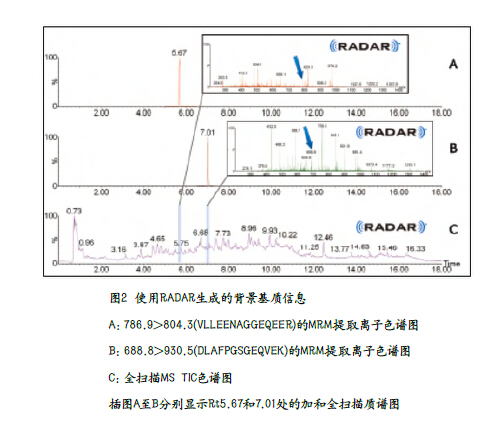
同时采集全扫描数据以及MRM数据能够在定量实验中评估基质的复杂程度。图2为全扫描总离子流图(TIC)以及每种肽的MRM色谱图。在每种肽的保留时间,其加和质谱图如插图所示。
如RADAR质谱图和TIC色谱图所示,消化后面包基质非常复杂,许多组分的色谱洗脱点很分散。这两种肽的各自离子信号都在质谱图中用箭头进行了标记,即使对于如此复杂的基质,肽的MRM提取离子色谱图仍然显示出优异的信噪比,说明Xevo TQ-S具有卓越的灵敏度和选择性。
总结
开发出一种快速和特异性的UPLC/MS/MS方法,能够使用ACQUITY UPLC和Xevo TQ-S检测食品基质中的过敏原肽标记,使用RADAR功能能够同时对化合物定量,用全扫描监测非常复杂的基质背景,使当前的方法扩展为一种多过敏原筛查方法变为可能。

[责任编辑:]

相关阅读
- (2017-05-11)白酒生产和贮藏过程中邻苯二甲酸酯类物质迁移研究
- (2018-01-04)标准物质使用中常见问答知识汇总!
- (2014-06-18)食品追溯体系保障“舌尖上的安全”
- (2014-06-18)沪8大类食品拟纳入追溯管理体系 或公示溯源信息
- (2014-06-18)伊利入选食品冷链物流管理首批试点企业
 Mettler-Toledo 在中国国际渔业博览会上展示创新的产品
Mettler-Toledo 在中国国际渔业博览会上展示创新的产品
 食品异物问题频发?是时候了解X射线检测了
食品异物问题频发?是时候了解X射线检测了
 开拓科技创新,撬动橡塑业高质量发展
开拓科技创新,撬动橡塑业高质量发展
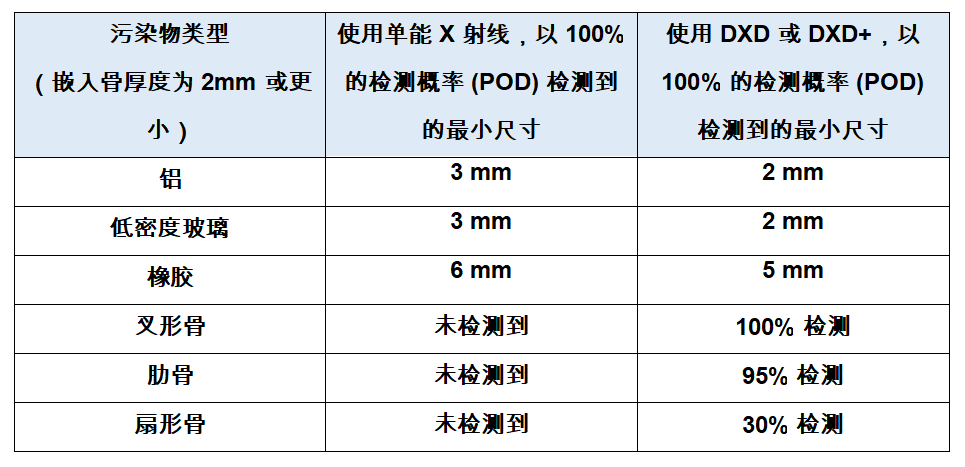 梅特勒托利多针对“难以发现”的污染物推出高品质X射线检
梅特勒托利多针对“难以发现”的污染物推出高品质X射线检
 探索婴幼儿辅食市场高质量发展之路,为宝宝成长保驾护航
探索婴幼儿辅食市场高质量发展之路,为宝宝成长保驾护航
 《食品安全最佳实践白皮书(2021-2022年)》四大主题发布
《食品安全最佳实践白皮书(2021-2022年)》四大主题发布
 《保健食品真实世界研究通则》团标技术审查与特食跨
《保健食品真实世界研究通则》团标技术审查与特食跨
 凝聚全球食饮智慧 SIAL西雅展国际化水平再创新高
凝聚全球食饮智慧 SIAL西雅展国际化水平再创新高
 精准把控 高质发展,第三届微生物安全与应用会议在
精准把控 高质发展,第三届微生物安全与应用会议在
 《食品行业科技创新白皮书》重磅发布!
《食品行业科技创新白皮书》重磅发布!






参与评论